Combien de sites ecori y a-t-il dans pbr322 ?
trois
Par la suite, on peut aussi se demander quel est le site de restriction de l’écor1 ?
En biologie moléculaire, il est utilisé comme une enzyme de restriction . EcoRI crée des extrémités collantes de 4 nucléotides avec des surplombs d’extrémité 5′ de AATT. La séquence de reconnaissance de l’acide nucléique dans laquelle l’ enzyme coupe est G/AATTC, qui a une séquence complémentaire palindromique de CTTAA/G.
Par ailleurs, combien de sites de restriction ont les plasmides ? Une fois qu’ils sont joints par la ligase, les fragments deviennent un seul morceau d’ADN intact. Le gène cible a maintenant été inséré dans le plasmide , ce qui donne un plasmide recombinant. Dans le plasmide , le gène est maintenant flanqué de deux sites EcoRI qui ont été générés lorsque les extrémités coupées ont été ligaturées ensemble.
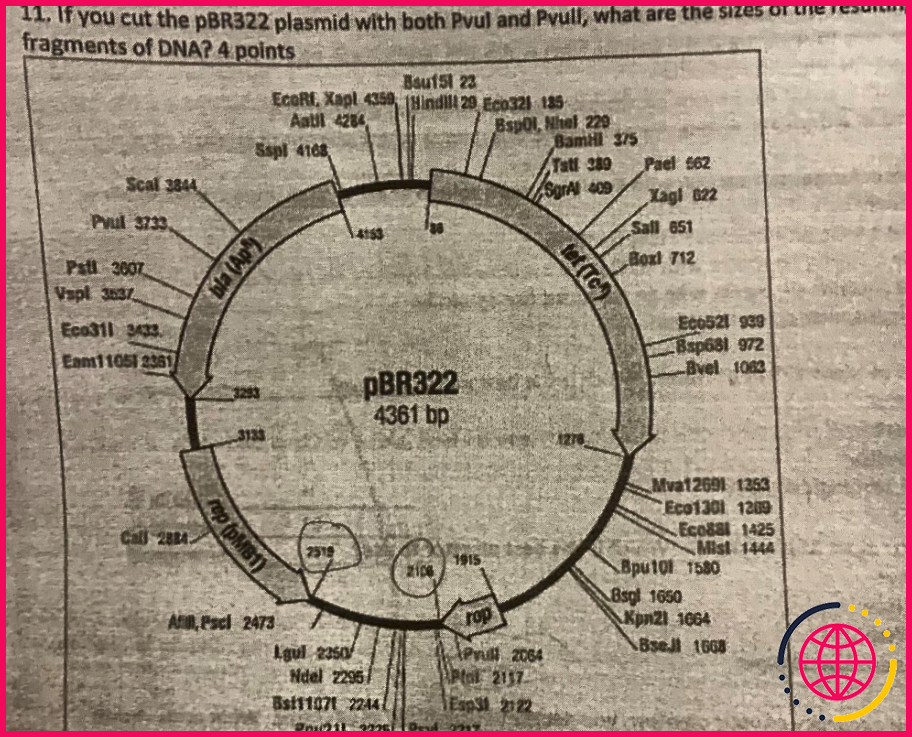
Par la présente, combien de paires de bases sont dans pBR322?
4,361
Que voulez-vous dire par pBR322?
pBR322 est un plasmide et a été l’un des premiers vecteurs de clonage E. coli largement utilisés. Le p signifie » plasmide « , et BR pour » Bolivar » et » Rodriguez « .
Quelle est la forme complète de EcoRI ?
EcoRI (prononcé ‘eco R one’) est une enzyme endonucléase de restriction isolée de l’espèce E. coli. EcoRI est une enzyme de restriction qui clive les doubles hélices d’ADN en fragments à des sites spécifiques. Elle fait également partie du système de modification de la restriction. En biologie moléculaire, elle est utilisée comme une enzyme de restriction.
Que signifie HindIII ?
HindIII (prononcé ‘Hin D Three’) est une enzyme de restriction désoxyribonucléasique de type II spécifique d’un site, isolée de Haemophilus influenzae, qui clive la séquence palindromique de l’ADN AAGCTT en présence du cofacteur Mg2+ par hydrolyse.
Quelle est la différence entre EcoRI et HindIII ?
Expliquez votre réponse. Les deux enzymes de restriction reconnaissent une séquence de six paires de bases, donc on s’attendrait à ce que les deux aient approximativement le même nombre de sites de reconnaissance par génome. La principale différence entre les deux est que EcoRI laisse des extrémités décalées, alors que SmaI laisse des extrémités émoussées.
Comment nomme-t-on une enzyme de restriction ?
Chaque enzyme est nommée d’après la bactérie dont elle a été isolée, en utilisant un système de dénomination basé sur le genre, l’espèce et la souche bactérienne. Par exemple, le nom de l’enzyme de restriction EcoRI a été dérivé comme indiqué dans l’encadré.
Qui a découvert l’ecor1 ?
Pour leur découverte des endonucléases de restriction (souvent appelées par le nom plus court d’enzymes de restriction) en 1970, Werner Arber, Hamilton Smith et Daniel Nathans ont reçu le prix Nobel de physiologie ou de médecine en 1978.
Que signifie le clonage de gènes ?
Le clonage de gènes est le processus dans lequel un gène d’intérêt est localisé et copié ( cloné ) à partir de l’ADN extrait d’un organisme. Lorsque l’ADN est extrait d’un organisme, tous ses gènes sont extraits en une seule fois. Cet ADN, qui contient des milliers de gènes différents.
Que fait BamHI ?
BamHI ( de BAcillus amyloli) est une enzyme de restriction de type II qui dérive de B. amyloliquefaciens , ayant la capacité de reconnaître de courtes séquences d’ADN et de les cliver spécifiquement à un site cible.
L’HindIII crée-t-elle des extrémités collantes ?
Couper l’ADN avec des enzymes de restriction peut produire des fragments avec des extrémités soit blondes soit collantes . HindIII – reconnaît la séquence 5’AAGCTT’3 – fins sèches . PstI – reconnaît la séquence 5’CTGCAG’3 – sticky ends . Sau3A – reconnaît la séquence 5’GATC’3 (produit les mêmes extrémités collantes que BamHI lors de la coupe)
.
Que signifie 322 dans pBR322 ?
Dans pBR322 , 322 représente le numéro attribué pour le ségréguer des autres types de plasmides. ou. il signifie l’ordre de synthèse.
Qu’est-ce que le plasmide pUC19 ?
pUC19 est un petit vecteur de clonage E. coli plasmide à nombre de copies élevé, dont les sites de clonage multiples comme indiqué ci-dessous. La molécule est un petit cercle à double brin, d’une longueur de 2686 paires de bases. La sélection du plasmide dans E. coli est conférée par le gène de résistance à l’ampicilline.
Le pBR322 est-il un vecteur d’expression ?
Le plasmide vecteur pBR322 est un vecteur de clonage polyvalent bien établi dans les laboratoires du monde entier, et un grand nombre de dérivés ont été créés pour des applications spécifiques et des objectifs de recherche, y compris l’ expression de gènes dans son hôte naturel, E. coli, et quelques autres bactéries.
Qu’est-ce qu’un gène marqueur sélectionnable ?
Un marqueur sélectionnable est un gène de sélection .
Gène introduit dans une cellule, notamment une bactérie ou à des cellules en culture, qui confère un caractère adapté à une sélection artificielle.
Qu’est-ce qu’un plasmide navette ?
Un vecteur shuttle est un vecteur (généralement un plasmide ) construit de manière à pouvoir se propager dans deux espèces hôtes différentes [1]. Par conséquent, l’ADN inséré dans un vecteur shuttle peut être testé ou manipulé dans deux types de cellules différentes.
Qu’est-ce que le plasmide ROP ?
Rop (également connu sous le nom de répresseur d’amorce) est une petite protéine responsable du maintien du nombre de copies de ColE1 et des plasmides bactériens apparentés à un faible niveau dans E. coli. Structurellement, Rop est une protéine homodimérique à faisceau de quatre hélices formée par l’interaction antiparallèle de deux monomères hélice-tour-hélice.
Qu’est-ce qu’un site de clonage multiple dans un plasmide ?
Un site de clonage multiple (MCS, ou région Polylinker) est une région d’ADN au sein d’un Plasmide qui contient des multiples sites uniques de coupure par enzyme de restriction. Les Plasmides sont très utiles en biotechnologie et une caractéristique clé de leur utilisation est le multiple site de clonage , qui permet d’insérer de l’ADN étranger dans le plasmide .
Qu’est-ce qu’un vecteur plasmidique ?
Un plasmide est une petite molécule d’ADN circulaire à double brin, distincte de l’ADN chromosomique d’une cellule. Les Plasmides qui sont utilisés expérimentalement à ces fins sont appelés vecteurs . Les chercheurs peuvent insérer des fragments d’ADN ou des gènes dans un plasmide vecteur , créant ainsi un plasmide recombinant.
Que sont les sites de clonage dans un vecteur de clonage ?
Site de clonage Tous les vecteurs de clonage ont des caractéristiques qui permettent d’insérer commodément un gène dans le vecteur ou de l’en retirer. Il peut s’agir d’un site de clonage multiple (MCS) ou d’un polylinker , qui contient de nombreux sites de restriction uniques.